Teste modelo de Engenharia Genética
1- Nomeie e
descreva de forma sumária os três principais passos da extracção de DNA genómico
de tecidos de mamíferos (não é necessário identificar reagentes químicos
específicos).
2- As enzimas de
restrição EcoRI e SmaI reconhecem as sequências descritas no quadro seguinte.
a) Se fosse possível
escolher qual delas utilizaria em clonagem? Justifique a resposta.
b) Em que
circunstâncias utilizaria a enzima preterida na alínea anterior?
|
EcoRI
|
5’-G’AATTC-3’
3’-CTTAA’G-5’
|
| |
|
|
SmaI
|
5’-CCC’GGG-3’
3’-GGG’CCC-5’
|
3- Para um
determinado gene foi possível isolar um clone de cDNA e outro de DNA genómico.
A partir dos dados fornecidos nas figuras seguintes é possível determinar
algumas características estruturais do gene e respectivo transcrito.
a) O
fragmento de cDNA foi excisado do plasmídeo com EcoRI e marcado com 32P por
“end labelling”. Após digestão simultânea com HaeIII e TaqI obtiveram-se
os seguintes fragmentos:

Após digestão
com TaqI obtiveram-se os seguintes fragmentos:
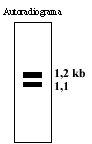
Determine o
mapa de restrição do cDNA.
b) O fragmento de
DNA genómico foi excisado dum clone de fago l com EcoRI. Após marcação com
32P por “end labelling” e digestão completa com BamHI obtiveram-se os
seguintes fragmentos:
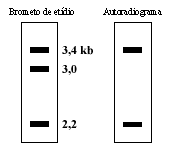
Determine o
mapa de restrição do DNA genómico
c)
Verificou-se que uma sonda de cDNA híbrida com os fragmentos de DNA genómico
de 3,4 e 2,2 kb. O fragmento de 1,2 kb da TaqI híbrida com o fragmento de DNA
genómico de 3,4 kb. Com qual fragmento de DNA genómico será de esperar que
hibride o fragmento de 0,5 kb da TaqI e HaeIII?
d) A que
parte estrutural do gene corresponde o fragmento de DNA genómico de 3,0 kb?
4- Descreva,
sumariamente, o método de marcação de DNA por “Nick translation”. Se
preferir pode usar esquemas.
5- Compare
(discutindo vantagens e desvantagens) o PCR (Polymerase chain reaction) e
clonagem como métodos de amplificação de DNA.
6- O método de
complementação alfa permite a identificação de clones de bactérias
recombinantes em função da capacidade das bactérias de degradar, ou não,
Xgal (substrato da b-galactosidase). Discuta a necessidade de utilização de um
sistema hospedeiro-vector específico para esta metodologia.
7- Discuta o binómio
restrição-metilação em clonagem.
8- Descreva um método
de screening de clones recombinantes.
9- Descreva, de
forma sucinta, e compare as técnicas de HA (Heteroduplex analysis) e SSCP
(Single strand conformation polymorphism) para detecção de mutações.
10- Imagine que
está a usar a E. coli para produzir uma enzima e descobre que a estabilidade térmica
desta é má. Um colaborador sugere-lhe que a substituição do aminoácido
isoleucina na posição 12 por uma cisteína irá melhorar a estabilidade térmica
desta. Descreva uma abordagem técnica que permita efectuar esta alteração.
11- Descreva e
discuta a importância da utilização de híbridos de células somáticas em
mapeamento genético tendo em conta a resolução desta técnica.
12- Discuta os
problemas potencialmente associados à produção de uma proteína humana num
sistema hospedeiro-vector procariótico.
13- Descreva, de
forma sumária, o estabelecimento de ratinhos KO.
14- Discuta a
seguinte frase: “A engenharia genética iniciou-se com a descoberta das
enzimas de restrição”.
