Pesquisa e identificação de mutações no gene da Caderina-E.
Num dos trabalhos práticos anteriores (estudo da expressão do gene da caderina-E por RT-PCR) foi descrita a função da caderina-E e foi discutido o seu envolvimento no desenvolvimento e progressão de vários tipos de tumores, nomeadamente no carcinoma difuso do estômago e no carcinoma lobular da mama.
Subjacentes às alterações de transcrição (estudadas no trabalho nº3) e às alterações estruturais da proteína estão mutações do gene da caderina-E
[1]. A inactivação, por mutação, deste gene está amplamente demonstrada em carcinomas difusos do estômago
[2] (Fig. 1).
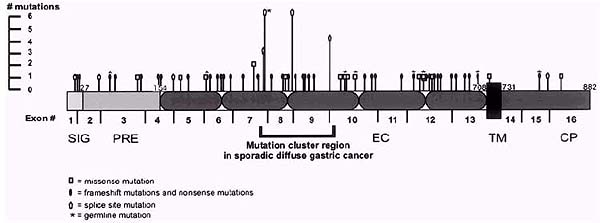
Fig. 1 – Distribuição, tipo e frequência de mutações da caderina-E em tumores e linhas celulares tumorais de origem humana. SIG, peptídeo de sinalização; PRE, sequência precursora; EC, domínios extracelulares; TM, domínio transmembranar; CP, domínio
citoplasmático.
O objectivo deste trabalho é pesquisar e identificar mutações do gene da caderina-E em carcinomas difusos do estômago usando a técnica de PCR-SSCP. A técnica de SSCP (Single Strand Conformation Polymorphism) permite identificar variações de sequência (mutações ou polimorfismos) através da detecção de alterações de conformação de cadeias simples de DNA; estas alterações de conformação são induzidas pelas variações de sequência.
Neste trabalho serão utilizados quatro amostras previamente submetidas a PCR (Fig. 2). Em cada uma das amostras foi amplificado um exão diferente do gene da caderina-E usando primers intrónicos flanqueadores dos exões (Tabela 1).
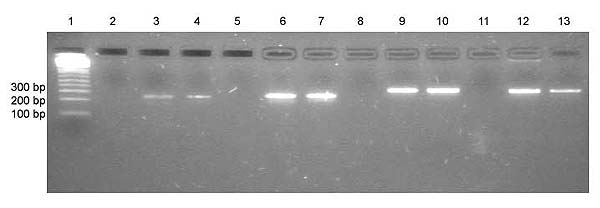
Fig. 2: Amplificação por PCR dos exões 4, 5, 7 e 10 do gene da caderina-E: 1- Marcador de peso molecular; 2, 3 e 4- Exão 4; 5, 6 e 7- Exão 5; 8, 9 e 10- Exão 7; 11, 12 e 13- Exão 10. 2, 5, 8 e 11- Controle negativo; 3, 6, 9 e 12- Amostra; 4, 7, 10 e 13- Controle normal.
Tabela 1 – Exão da caderina-E amplificado em cada uma das quatro amostras e respectivo tamanho do produto de
PCR.
|
Amostra |
Exão |
Amplicon (bp) |
|
1 |
4 |
237 |
|
2 |
5 |
256 |
|
3 |
7 |
328 |
|
4 |
10 |
308 |
A sequência “wild-type” para cada um dos produtos de PCR é a seguinte (sublinhado, primer; bold, exão).
Amplicon 4
CTTGTTCCTCATCTTCTTTCCTTTTAGGCCTCCGTTTCTGGAATCCAAGCAGAATTGCTCACATTT
CCCAACTCCTCTCCTGGCCTCAGAAGACAGAAGAGAGACTGGGTTATTCCTCCCATCAGCTGCCCA
GAAAATGAAAAAGGCCCATTTCCTAAAAACCTGGTTCAGGTAGAGAAAGAAGTTCTCTGTTTCTCT
GGGAGGGATTTGGCAGAGAAGTACCAAGGAGAGAAAGGG
Amplicon 5
GTTGGGATCCTTCTTTACTAATTCTTTTTCTTTCATTTTGTCTTCAGATCAAATCCAACAAAGACA
AAGAAGGCAAGGTTTTCTACAGCATCACTGGCCAAGGAGCTGACACACCCCCTGTTGGTGTCTTTA
TTATTGAAAGAGAAACAGGATGGCTGAAGGTGACAGAGCCTCTGGATAGAGAACGCATTGCCACAT
ACACTGTAAGTATCTCTTAGAAGCTTGTTGACACCGGGGTAACATCCACCCAGGATTT
Amplicon 7
AGCTTGTCTAAACCTTCATCTCCTTGAACTCTTCCAGGAACCTCTGTGATGGAGGTCACAGCCACA
GACGCGGACGATGATGTGAACACCTACAATGCCGCCATCGCTTACACCATCCTCAGCCAAGATCCT
GAGCTCCCTGACAAAAATATGTTCACCATTAACAGGAACACAGGAGTCATCAGTGTGGTCACCACT
GGGCTGGACCGAGAGGTCAGGGGTCAGGAGGATCCAGAGGGTGTGGAGGACAAATGTGTATTAGCT
CAATCCCGTGGACAAGCAAAATCCTGCTAGGCCAGTTGTCAGTTAATACAGTGATGGTCTAAGC
Amplicon 10
ACTTCATTGTTTCTGCTCTCTAGGGCTTGGATTTTGAGGCCAAGCAGCAGTACATTCTAACGTAGC
AGTGACGAATGTGGTACCTTTTGAGGTCTCTCTCACCACCTCCACAGCCACCGTCACCGTGGATGT
GCTGGATGTGAATGAAGCCCCCATCTTTGTGCCTCCTGAAAAGAGAGTGGAAGTGTCCGAGGACTT
TGGCGTGGGCCAGGAAATCACATCCTACACTGCCCAGGAGCCAGACACATTTATGGAACAGAAAAT
AACGTAAGTGTGAGGATTTTTCAACTGACTTGCAGCAACTGGTT
Numa das próximas aulas práticas (visita de estudo ao IPATIMUP) serão obtidas as sequenciações dos variantes de SSCP obtidos no presente trabalho. As sequências “wild-type” servirão de padrão para analisar as
sequenciações.
O presente trabalho, em conjunto com os resultados da sequenciação, implica a elaboração de um relatório científico que deverá obedecer ao formato clássico deste tipo de textos: título e identificação do autor; introdução (disponibilizando a informação mínima necessária para compreender a pergunta que vai ser feita); material e métodos (listagem de material e explicação sucinta da metodologia); resultados (descrição factual dos resultados, sempre que possível com recurso a imagens, esquemas e tabelas); discussão (interpretação dos resultados, estabelecimento de hipóteses explicativas e conclusões); bibliografia (usar o formato da bibliografia deste texto - última página).
Como sugestão para a discussão do relatório fornecem-se as seguintes palavras-chave: caderina-E; mutação; polimorfismo; tipo de mutação; carcinoma difuso do estômago.
Protocolo
ATENÇÃO:
Alguns dos produtos químicos usados neste trabalho são bastante tóxicos. Manipular o formaldeído e ácido acético na hote. Evitar o contacto com nitrato de prata e MDE.
I- Gel de MDE
Para cada exão serão analisadas duas amostras: a amostra em estudo e um controle normal. Para controlo da desnaturação das amostras será incluída uma amostra não desnaturada para cada
exão, o que perfaz um total de 12 amostras.
1- Montar o sistema de electroforese vertical.
- Lavar cuidadosamente os vidros do sistema, spacers e pentes com água e etanol 70%.
- Secar bem os diferentes componentes.
- Montar os vidros e spacers nos grampos e no sistema de preparação do gel.
2- Preparar o gel.
- Para cada gel misturar:
- 14 ml 2xMDE
- 1,75 ml 20xTBE
- 19,25 ml Água desionizada
- 35 ml TEMED
- 270 ml AMPS 10%
- Imediatamente após a adição de AMPS verter o gel no respectivo sistema de preparação com a ajuda de uma seringa de 20 ml ou mais.
- Inserir o pente no gel.
- Esperar 1 hora antes de utilizar o gel e verificar o sucesso da polimerização na solução excedente.
3- Electroforese.
- Retirar os géis do sistema de preparação e montá-los no sistema de electroforese.
- Colocar 1xTBE no sistema de electroforese.
- Desnaturar as amostras a 950C durante 10 mins.
- Colocar as amostras rapidamente em gelo durante pelo menos 1 minuto antes de iniciar o carregamento do gel.
- Manter as amostras em gelo e carregar 8 ml de amostra em cada poço do gel.
- Colocar os géis no tanque de electroforese e completar o volume com 1xTBE.
- Correr a electroforese a 170V, 80C durante cerca de 15 horas (xileno cianol @ a 2/3 do gel).
II- Coloração do gel
1- Preparar as soluções de fixação, nitrato de prata (ver em baixo passo 3) e revelação (sem juntar formaldeído).
2- Cuidadosamente desmontar o sistema de electroforese, abrir os vidros, marcar a orientação dos géis e colocar os géis em 480 ml de solução fixadora (com agitação) durante 10 mins.
3- Dissolver 1g de nitrato de prata em 20 ml de solução fixadora e adicionar à solução anterior. Agitar durante 10 mins.
3- Lavar bem em água desionizada três vezes.
4- Adicionar 500 ml de formaldeído à solução de revelação. Adicionar cerca de 30 ml de revelador aos géis e lavar rapidamente para remover excesso de nitrato de prata. Adicionar o resto do revelador e agitar até as bandas aparecerem.
5- Colocar o gel em novo fixador durante 5 mins.
6- Passar os géis para papel 3MM, cobrir com película aderente e secar a 650C durante 1 hora.
Bibliografia
1- Berx G, Becker KF, Hofler H, van Roy F. Mutations of the human E-cadherin (CDH1) gene. Hum Mutat 12: 226-237, 1998.
2- Machado JC, Soares P, Carneiro F, Rocha A, Beck S, Blin N, Berx G, Sobrinho-Simões M. E-cadherin gene mutations provide a genetic basis for the phenotypic divergence of mixed gastric carcinomas. Lab Invest 79: 459-465, 1999.
